Núcleo celular
| Biologia celular |
|---|
| Célula animal |
Organelos da célula animal 
|
| Célula vegetal |
Organelos da célula vegetal 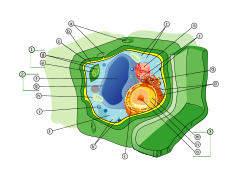
1. Cloroplasto
2. Vacúolo
3. Núcleo
|
Portal Biologia celular Portal Biologia |

Figura do núcleo e do retículo endoplasmático: (1) Envoltório nuclear. (2) Ribossomos. (3) Poros nucleares. (4) Nucléolo. (5) Cromatina. (6) Núcleo. (7) Retículo endoplasmático. (8) Nucleoplasma.
O núcleo celular, primeiramente descrito por Franz Bauer, em 1802, é uma estrutura presente nas células eucariontes, que contém o ADN (ou DNA) da célula. É delimitado pelo envoltório nuclear, e se comunica com o citoplasma através dos poros nucleares. O núcleo possui duas funções básicas: regular as reações químicas que ocorrem dentro da célula (metabolismo), e armazenar as informações genéticas da célula. O seu diâmetro pode variar de 10 a 22.25 μm.
Além do material genético, o núcleo também possui algumas proteínas com a função de regular a expressão gênica, que envolve processos complexos de transcrição, pré-processamento do mRNA (RNA mensageiro), e o transporte do mRNA formado para o citoplasma. Dentro do núcleo ainda se encontra uma estrutura denominada nucléolo, que é responsável pela produção de subunidades dos ribossomos. O envoltório nuclear é responsável tanto por separar as reações químicas que ocorrem dentro do citoplasma daquelas que ocorrem dentro do núcleo, quanto por permitir a comunicação entre esses dois ambientes. Essa comunicação é realizada pelos poros nucleares que se formam da fusão entre a membrana interna e a externa do envoltório nuclear.
O interior do núcleo é composto por uma matriz denominada de nucleoplasma, que é um líquido de consistência gelatinosa, similar ao citoplasma. Dentro dele estão presentes várias substâncias necessárias para o funcionamento do núcleo, incluindo bases nitrogenadas, enzimas, proteínas e fatores de transcrição. Também existe uma rede de fibras dentro do nucleoplasma (chamada de matriz nuclear), cuja função ainda está sendo discutida.
O ADN presente no núcleo encontra-se geralmente organizado na forma de cromatina (que pode ser eucromatina ou heterocromatina), durante o período de interfase. Durante a divisão celular, porém, o material genético é organizado na forma de cromossomos. Sua posição é geralmente central, acompanhando o formato da célula, mas isso pode variar de uma para outra. Nos eritrócitos dos mamíferos, o núcleo está ausente.
Índice
1 História
2 Estrutura do núcleo celular
2.1 Citoesqueleto
2.2 Cromossomos
2.3 Envelope nuclear e poros nucleares
2.4 Nucléolo
2.5 Outros corpos subnucleares
2.5.1 Corpos de Cajal e gémeos
2.5.2 Domínios PIKA e PTF
2.5.3 Corpos PML
2.5.4 Paraspeckles
2.5.5 Agregados granulares intercromatínicos
3 Função
3.1 Compartimentação celular
3.2 Expressão genética
3.3 Processamento do pré-ARNm
4 Dinâmica e regulação
4.1 Transporte nuclear
4.2 Agregação e desagregação
5 Células anucleadas e polinucleadas
6 Evolução
7 Referências
8 Leitura adicional
9 Ligações externas
História |

Desenho de um núcleo celular, por Walther Flemming, feito em 1882.
O núcleo celular foi o primeiro organelo a ser descoberto, tendo sido primeiramente descrito por Franz Bauer, em 1802.[1] Foi mais tarde descrito em mais detalhe pelo botânico escocês Robert Brown, em 1831, numa palestra na Sociedade Linneana de Londres. Brown estava a estudar orquídeas ao microscópio quando observou uma região opaca, que chamou de auréola ou núcleo, existentes nas células da camada exterior, em flores.[2] Na altura não sugeriu nenhuma potencial função. Em 1838, Matthias Schleiden propôs que o núcleo desempenhava um papel na geração de células, tendo introduzido o nome "citoblasto" (gerador de células). Acreditou que tinha observado novas células a aparecerem à volta dos "citoblastos". Franz Meyen era um forte opositor a esta teoria, tendo já descrito células a multiplicar-se por divisão e acreditando que muitas células não teriam núcleo. A ideia de que as células podem ser geradas de novo, pelo "citoblasto", contradizia os trabalhos de Robert Remak (1852) e Rudolf Virchow (1855), que decisivamente propagaram o paradigma de que as células são geradas somente por outras células ("Omnis cellula e cellula"). A função do núcleo permanecia, no entanto, pouco clara.[3]
Entre 1876 e 1878, Oscar Hertwig publicou vários estudos sobre a fertilização em óvulos de ouriço-do-mar, mostrando que o núcleo do espermatozoide entra no oócito, fundindo-se com o seu núcleo. Esta foi a primeira vez que era sugerido que um indivíduo se desenvolve a partir de uma única célula nucleada. Isto vinha em contradição com a teoria de Ernst Haeckel, de que a filogenia completa de uma espécie era repetida durante o desenvolvimento embrionário, incluindo a geração da primeira célula nucleada a partir de uma "Monerula", uma massa sem estrutura, de muco primordial ("Urschleim"). A necessidade de um núcleo espermático para a fertilização foi discutida por algum tempo. No entanto, Hertwig confirmou as suas observações em outros grupos animais, como por exemplo em anfíbios e moluscos. Eduard Strasburger produziu os mesmos resultados em plantas (1884). Isto abriu o caminho para estabelecer o núcleo como tendo um papel primordial na hereditariedade. Em 1873, August Weismann postulou a equivalência das células germinais paternais e maternas para a hereditariedade. A função do núcleo, como transportador da informação genética, apenas ficou clara mais tarde, após a mitose ter sido descoberta e a hereditariedade mendeliana ter sido redescoberta, no início do século XX. Nessa altura, a teoria cromossómica da hereditariedade foi desenvolvida.[3]
Estrutura do núcleo celular |
O núcleo é o maior organelo celular em animais.[4] Em células de mamíferos, o diâmetro médio anda tipicamente à volta de 11 a 22μm e ocupa 10% do volume total.[5] O líquido viscoso dentro do núcleo denomina-se nucleoplasma, e é similar ao citoplasma encontrado no exterior do núcleo.
Citoesqueleto |
Nas células animais, duas redes de filamentos intermédios providenciam suporte estrutural ao núcleo: a lâmina nuclear forma uma rede organizada na face interna do envelope, enquanto que um tipo de suporte menos organizado é providenciado pela face citosólica do envelope. Ambos os sistemas dão o suporte estrutural para o envelope nuclear e atuam como pontos de ancoragem para os cromossomas e poros nucleares.[5]
A lâmina nuclear é essencialmente composta por proteínas denominadas laminas. Como todas as proteínas, as laminas são sintetizadas no citoplasma e depois transportadas para o interior do núcleo, onde são agregadas antes de serem incorporadas na rede existente de lâmina nuclear.[6][7] As laminas podem também ser encontradas dentro do nucleoplasma, onde formam uma estrutura regular[8] que é visível com o auxílio de microscopia de fluorescência. A função desta estrutura ainda não está totalmente estabelecida, embora se saiba que está excluída do nucléolo e está presente durante a interfase.[9] As estruturas de laminas que formam esta estrutura ligam-se à cromatina e rompendo a sua estrutura dá-se a inibição da transcrição de genes que codificam proteínas.[10]
Tal como os componentes de outros filamentos intermédios, o monómero de lamina contém um domínio em alfa-hélice, usados por dois monómeros para se enrolarem um no outro, formando uma estrutura dimérica denominada coiled-coil. Então, duas destas estruturas diméricas colocam-se lado a lado, num arranjo antiparalelo, formando um tetrâmero denominado protofilamento. Oito destes protofilamentos formam um arranjo lateral que é torcido de molde a formar uma estrutura semelhante a uma corda. Estes filamentos podem ser juntos ou separados de uma maneira dinâmica, significando que o comprimento do filamento depende das diferentes taxas de adição e remoção de filamento.[5]
Cromossomos |
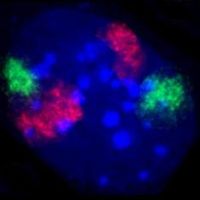
O núcleo de um fibroblasto de um rato, no qual o ADN está colocado do azul. Os distintos territórios cromossómicos, do cromossoma 2 (a vermelho) e do cromossoma 9 (a verde) são visíveis através de coloração hibridização fluorescente in situ.
O núcleo celular contém a maioria do material genético da célula, sob a forma de múltiplas moléculas lineares de ADN organizadas em estruturas denominadas cromossomos. Durante a maior parte do ciclo celular estão organizados num complexo ADN-proteína conhecido como cromatina, e durante a divisão celular a cromatina pode ser vista a formar os cromossomas bem definidos que são familiares de um cariótipo. Uma pequena fração dos genes da célula está localizada na mitocôndria.
Existem dois tipos de cromatina. A eucromatina é a forma menos compacta de ADN, e contém genes que são frequentemente expressos pela célula.[11] O outro tipo, a heterocromatina, é a forma mais compacta, e contém ADN que não é frequentemente transcrito. Esta estrutura é ainda mais categorizada em heterocromatina facultativa, consistindo de genes que estão organizados como heterocromatina apenas em certos tipos de célula ou em certos estágios de desenvolvimento, e a heterocromatina constitutiva, que consiste em componente cromossómicos estruturais como os telómeros e os centrómeros.[12] Durante a interfase, a cromatina organiza-se em pequenos aglomerados individuais,[13] denominados territórios cromossómicos.[14] Os genes ativos, que são normalmente encontrados na região da eucromatina, tendem a estar localizados nas fronteiras deste territórios cromossómicos.[15]
Anticorpos associados com certos tipos de organização da cromatina, particularmente os nucleossomas, têm sido relacionados com um número de doenças autoimunes, tal como o lupus eritematoso sistémico.[16] Estes são conhecidos como anticorpos antinucleares (AAN) e têm sido observados concertadamente com esclerose múltipla, como parte de uma disfunção geral do sistema imunitário.[17]
Envelope nuclear e poros nucleares |
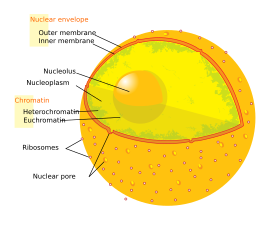
O núcleo da célula eucariota. Neste diagrama é visível a dupla membrana do envelope nuclear, impregnada de ribossomas, o ADN e o nucléolo. Dentro do núcleo existe um líquido viscoso denominado nucleoplasma, similar ao do citoplasma que se encontra fora do núcleo.
O invólucro nuclear é composto por duas membranas celulares dispostas em paralelo (uma interior e outra exterior) e separadas por 10 a 50 nanómetros. O envelope nuclear envolve completamente o núcleo e separa o material genético da célula do citoplasma, servindo como barreira à difusão livre de macromoléculas entre o nucleoplasma e o citoplasma[18] A membrana nuclear externa é contínua com a membrana do retículo endoplasmático rugoso (RER), estando igualmente recoberta de ribossomas. O espaço entre as membranas nucleares é chamado de espaço perinuclear e tem continuidade com o lúmen do RER.
Os poros nucleares providenciam canais aquosos através do invólucro, sendo compostos por múltiplas proteínas, colectivamente denominadas de nucleoporinas. Os poros possuem cerca de 125 milhões de dalton de massa molecular e consistem em cerca de 50 (em leveduras) a 100 proteínas (em vertebrados).[4] Os poros possuem 100 nm de diâmetro total; no entanto, o espaço através do qual as substâncias difundem livremente tem apenas 9 nm de largura, devido à presença de sistemas de regulação no centro do poro. Este tamanho permite a livre passagem de pequenas moléculas solúveis em água ao mesmo tempo em que impede que moléculas de maiores dimensões, como os ácidos nucleicos e proteínas entrem ou saiam de maneira inapropriada. Estas moléculas maiores terão que ser transportadas para o interior do núcleo de maneira ativa. O núcleo de uma típica célula de mamífero tem cerca de 3000 a 4000 poros através de todo o seu envelope,[19] com cada um deles contendo uma estrutura anelar, de simetria octogonal, no local onde as membranas interna e externa se fundem.[20] Ligado a este anel existe uma estrutura em forma de cesto que se estende em direção ao nucleoplasma, e uma série de extensões filamentosas que alcançam o citoplasma. Ambas as estruturas servem para mediar a ligação a proteínas transportadoras nucleares.[4]
A maioria das proteínas, subunidades ribossomais e alguns ARN são transportados através dos complexos de poros num processo mediado por uma família de factores de transporte denominadas carioferinas. Estas carioferinas que medeiam o movimento para o núcleo também são chamadas de importinas, enquanto que aquelas que medeiam o movimento para fora do núcleo são chamadas de exportinas. A maioria das carioferinas interage diretamente com a sua carga, apesar de algumas usarem proteínas adaptadoras.[21]hormonas esteroides como o cortisol e a aldosterona, tal como outras pequenas moléculas lipossolúveis, envolvidas na sinalização intercelular, podem se difundir através da membrana celular, para o citoplasma, onde se ligam a receptores nucleares que são transportados para o núcleo. Já no núcleo, servem como factores de transcrição quando juntos com o seu ligando; na ausência do ligando, muitos receptores funcionam como desacetilases de histonas que reprimem a expressão genética.[4]
Nucléolo |

Micrografia electrónica de um núcleo celular, mostrando um nucléolo com uma coloração escura.
O nucléolo é uma estrutura presente dentro do núcleo, nâo envolta por membrana. Por vezes é classificado como suborganelo. Forma-se em volta de repetições de ADNr, ADN que codifica o ARN ribossomal (ARNr). Estas regiões são denominadas regiões organizadoras de nucléolo. O papel principal do nucléolo é o de sintetizar ARNr e de formar os ribossomas. A coesão estrutural do nucléolo depende da sua atividade, já que a formação de ribossomas resulta na associação temporária de componente nucleolares, facilitando assim mais formação de ribossomas e logo uma maior associação. Este modelo é suportado por observações de que a inativação do ADNr resulta na mistura de componentes nucleolares.[22]
O primeiro passo na formação do ribossoma é a transcrição do ADNr, efectuado por uma proteína chamada RNA polimerase I, dando origem a um pré-ARNr precursor, de grandes dimensões. Este é clivado nas subunidades 5.8S, 18S, e 28S do ARNr.[23] A transcrição, o processamento pós-transcricional e a formação do ribossoma, ocorrem no nucléolo, auxiliado por moléculas de ARN nucleolar pequeno (snoRNA, em inglês), algumas das quais derivado de splicing de intrões de genes codificantes de ARN mensageiro, relacionados com funções ribossomais. As subunidades ribossomais já formadas são as estruturas de maior dimensão que passam pelos poros nucleares.[4]
Quando observado através do microscópio electrónico, o nucléolo pode ser visto como sendo constituído por três regiões distintas: uma região interior (centro fibrilar), rodeada pelo componente fibrilar denso, que por sua vez é rodeado pelo componente granular. A transcrição do ADNr ocorre no centro fibrilar ou na fronteira entre o centro fibrilar e o componente fibrilar denso. Quando a transcrição de ADNr é aumentada, verifica-se a detecção de mais centros fibrilares. A maior parte da clivagem e modificação do ARNr ocorre no componente fibrilar denso, enquanto que os passos mais tardios, envolvendo a assemblagem de proteínas em subunidades ribossomais, ocorre no centro granular.[23]
Outros corpos subnucleares |
Nome da estrutura | Diâmetro da estrutura |
|---|---|
| Corpos de Cajal | 0.2–2.0 µm[24] |
| PIKA | 5 µm[25] |
| Corpos PML | 0.2–1.0 µm[26] |
| Paraspeckles | 0.2–1.0 µm[27] |
| Speckles | 20–25 nm[25] |
Para além do nucléolo, o núcleo contém um número de outros corpos não-membranares. Alguns deles são os corpos de Cajal, os gémeos de corpos enovelados (gemini of coiled bodies, em inglês). Domínios PIKA, corpos PML, agregados de grânulos intercromatínicos (speckles) e paraspeckles. Apesar de pouco se saber sobre alguns destes domínios, estes são significantes pelo facto de mostrarem que o nucleoplasma não é uniforme, mas sim que contém vários subdomínios funcionais organizados.[26]
Outras estruturas subnucleares aparecerem como parte de processos de doenças. Por exemplo, foi já reportada a presença de pequenos bastões intranucleares em alguns casos de miopatia nemalínica.
Corpos de Cajal e gémeos |
Um núcleo contém tipicamente entre uma a dez estruturas denominadas corpos de Cajal ou corpos enovelados, cujo diâmetro é de 0,2 µm e 2.0 µm, dependendo do tipo de célula e da espécie.[24] Quando vistos ao microscópio electrónico, assemelham-se a novelos[25] e são densos focos de distribuição para a proteína denominada coilina.[28] Estes corpos estão envolvidos em alguns papéis relacionados com o processamento do ARN, especificamente os pequenos ARN nucleolares (snoRNA), a maturação dos pequenos ARN nucleares (snRNA) e modificação do ARNm histónico.[24]
Similares aos corpos de Cajal são os gémeos de corpos enovelados, quer em forma quer em tamanho. São virtualmente indistinguíveis sob o microscópio electrónico.[28] Em oposição aos corpos de Cajal, os gémeos não possuem pequenas ribonucleoproteínas nucleares (snRNPs), mas contêm uma proteína em inglês denominada survivor of motor neurons (SMN), cuja função está relacionada com a biogénese das snRNP. Supõe-se que os gémeos assistem os corpos enovelados na biogénese das snRNP,[29] apesar de também ter sido sugerido, de evidências microscópicas, que os corpos enovelados os os gémeos de corpos enovelados são diferentes manifestações da mesma estrutura.[28]
Domínios PIKA e PTF |
Os domínios PIKA (do inglês, polymorphic interphase karyosomal associations) foram primeiramente descobertos em estudos de microscopia no ano 1991. As suas funções eram e permanecem pouco claras, apesar de não terem sido associados com replicação ativa de ADN, com a trasncrição e com o processamento do ARN.[30] Descobriu-se que se associavam com distintos domínios definidos por densas localizações do factor de transcrição PTF, que promove a transcrição de snRNA.[31]
Corpos PML |
Os corpos PML (do inglês, promyelocytic leukaemia) são corpos esféricos que se encontram dispersos por todo o nucleoplasma, medindo entre 0,2 e 1,0 µm. Outros nomes são: domínio nuclear 10, corpos Kremer e domínios oncogénicos PML. São muitas vezes vistos no núcleo em associação a corpos de Cajal e a corpos de clivagem. Foi sugerido que desempenham um papel na regulação da transcrição.[26]
Paraspeckles |
Descobertos por Fox et al. em 2002, os paraspeckles são compartimentos de forma irregular que ocorrem no espaço intercromatínico[32] Foram documentados pela primeira vez em células HeLa, onde existem em número de 10 a 30 por núcleo.[33] Também se conhece a sua ocorrência em células primárias humanas, em linhas celulares trnasformadas e em secções de tecidos.[34]
Os paraspeckles são estruturas dinâmicas que são alteradas em resposta a mudanças na atividade metabólica celular. São dependente de transcrição[32] e em ausência de transcrição por ARN Pol II estas estruturas desaparecem e todos os seus componentes proteicos associados (PSP1, p54nrb, PSP2, CFI(m)68 e PSF) formam uma estrutura em forma de crescente, em posição perinucleolar. Este fenómeno é demonstrado durante o ciclo celular. Durante o ciclo celular, os paraspeckles estão presentes durante a interfase e durante a toda a mitose, com excepção da telófase. Durante a telofase, quando os dois núcleos-filho são formados, não existe transcrição por ARN polimerase II, de tal forma que os componentes proteicos formam uma cobertura perinucleolar.[34]
Agregados granulares intercromatínicos |
Os agregados granulares intercromatínicos ou speckles (speckles de clivagem) são ricos em snRNPs de clivagem e em outras proteínas necessárias para o processamento do pré-ARNm. Porque a célula tem necessidades variáveis, a composição e a localização destes corpos muda em função da transcrição do ARNm e da regulação via fosforilação de proteínas específicas.[35]
Função |
A principal função do núcleo celular é controlar a expressão genética e mediar a replicação do ADN durante o ciclo celular. O núcleo providencia o local para a transcrição, que está separado do local da tradução, no citoplasma. Isto permite um nível de regulação genética que não está disponível nos procariotas.
Compartimentação celular |
O envelope nuclear permite que o núcleo controle o seu conteúdo, separando-o do resto do citoplasma quando necessário. Isto é importante para o controlo dos processos de ambos os lados da membrana nuclear. Em alguns casos, onde um processo citoplasmático necessita de ser restringido, um componente chave é removido para o núcleo, onde interage com factores de transcrição que regulam a produção de certas enzimas nas vias metabólicas. Este mecanismo regulador ocorre no caso da glicólise, uma via metabólica que age para degradar a glucose para produzir energia. A hexoquinase é uma enzima responsável pelo primeiro passo da glicólise, formando glucose-6-fosfato a partir da glucose. A altas concentrações de frutose-6-fosfato, uma proteína reguladora remove a hexoquinase para o núcleo,[36] onde forma onde complexo transcricional repressor juntamente com proteínas nucleares, para reduzir a expressão de genes envolvidos na glicólise.[37]
De maneira a controlar quais genes são transcritos, a célula impede que alguns factores de transcrição, responsáveis por regular a expressão genética, de terem acesso ao ADN, até que sejam ativados por outras vias de sinalização. Isto previne até mesmo níveis baixos de expressão genética inapropriada. Por exemplo, no caso de genes controlados por NF-κB, envolvidos na maioria das respostas inflamatórias, a transcrição é induzida em resposta a uma via de sinalização, como aquela que é iniciada pela molécula sinalizadora denominada TNF-α, que se liga a um receptor na membrana celular, resultando no recrutamento de proteínas sinalizadoras e eventualmente na ativação do factor de transcrição NF-κB. Um sinal de localização nuclear na proteína NF-κB, permite que seja transportada através do poro nuclear até ao núcleo, onde estimula a transcrição dos genes-alvo.[5]
A compartimentação permite que a célula previna a tradução de mRNA que não sofreu splicing.[38] O mRNA eucariota contém intrões que devem ser removidos antes que ocorra a tradução e dêem origem a proteínas funcionais. O splicing é efectuado dentro do núcleo antes de o mRNA poder ser acedido por ribossomas para se dar a tradução. Sem o núcleo, os ribossomas iriam traduzir o mRNA recentemente transcrito (não processado) em proteínas com malformações e não funcionais
Expressão genética |

Trascrição genética a decorrer.
A expressão genética envolve a transcrição, na qual o ADN é usado como modelo para a produção de ARN. No caso de genes que codificam proteínas, o ARN produzido por este processo é o ARN mensageiro, que depois necessita de ser traduzido pelos ribossomas para formação das proteínas. Como os ribossomas se localizam fora do núcleo, o ARNm produzido necessita de ser exportado.[39]
Uma vez que o núcleo é o local da transcrição, também contém uma variedade de proteínas que ou fazem a mediação direta da transcrição ou estão envolvidos em regular o processo. Estas proteínas incluem as helicases que desenrolam a dupla fita da molécula da ADN, para facilitar a acesso a ela, a ARN-polimerase que sintetiza a molécula de ARN, a topoisomerase que muda a quantidade de enrolamento no ADN, assim como uma grande variedade de factores de transcrição que regulam a expressão genética.[40]
Processamento do pré-ARNm |
As moléculas recém criadas de ARNm são conhecidas como transcritos primários. Elas têm que sofrer modificação pós-transcricional no núcleo antes de serem exportadas para o citoplasma; o ARNm que aparece no núcleo sem estas modificações é degradado em vez de traduzido em proteínas. As três principais modificações são: inserção de uma capa na extremidade 5', poliadenilação na extremidade 3', e splicing de ARN. Enquanto no núcleo, o pré-ARN está associado com uma variedade de proteínas, em complexos denominados partículas de ribonucleoproteínas heterogéneas (hnRPNs). A adição da capa 5´ocorre cotranscricionalmente e é o primeiro passo na modificação pós-transcricional]]. A cauda múltipla de adenina na extermidade 3' é apenas adicionada após a transcrição estar completa.
O splicing do ARN, levado a cabo por um complexo denominado spliceossoma, é o processo pelo qual os intrões, ou regiões do ADN que não codificam proteínas, são removidas do pré-ARNm e o remanescente exão é reconectado numa molécula contínua. Este processo normalmente ocorre após a inserção da capa 5' e da poliadenilação 3', mas pode ter início antes da síntese estar completa em transcritos com muitos exões.[4] Muitos pré-ARNm, incluindo aqueles que codificam anticorpos, podem sofrer splicing de variadas formas, produzindo diferentes ARMm maduros que codificam proteínas com diferentes estruturas primárias. Este processo é conhecido com splicing alternativo e permite a produção de uma grande variedade de proteínas a partir de uma quantidade limitada de ADN.
Dinâmica e regulação |
Transporte nuclear |

Macromoléculas, como o ARN e proteínas, são transportadas ativamente através da membrana nuclear, num processo denominado ciclo Ran-GTP de transporte nuclear.
A entrada e saída de grandes moléculas do núcleo está intimamente controlada pelos complexos de poros nucleares. Apesar de pequenas moléculas poderem entrar no núcleo sem regulação,[41] macromoléculas como o ARN e proteínas requerem associação com carioferinas denominadas importinas para entrar no núcleo e exportinas para sair. Proteínas de carga que têm que ser transferidas do citoplasma para o núcleo contêm sinais de localização nuclear ligadas pelas exportinas. A habilidade das importinas e exportinas em transportar a sua carga é regulada por GTPases, enzimas que hidrolisam a molécula de guanosina trifosfato para libertar energia. A GTPase de maior importância envolvida no transporte nuclear denomina-se Ran, que pode se ligar a GTP ou GDP, dependendo se estiver localizada no núcleo ou citoplasma. Enquanto que as importinas dependem de RanGTP para se dissociarem da sua carga, as exportinas requerem RanGTP para se poderem ligar à sua carga.[21]
A importação nuclear depende da importina se ligar à sua carga, no citoplasma, e transportá-la através do poro nuclear até ao núcleo. Dentro do núcleo, a RanGTP atua para separar a carga da importina, permitindo que esta possa sair do núcleo para ser reutilizada. A exportação nuclear é similar, sendo que a exportina liga-se à carga dentro do núcleo, num processo facilitado pela RanGTP, saindo depois através do poro nuclear, separando depois da sua carga no citoplasma.
Proteínas de exportação, especializadas, existem para efectuar a transferência de Arnm madura e ARNt para o citoplasma, após a modificação pós-transcripcional estar completa. Este mecanismo de controlo de qualidade é importante devido ao papel central destas moléculas no processo de tradução das proteínas; uma expressão errada de uma proteína devido à incompleta excisão de intrões ou a incorreta incomporação de aminoácidos, poderão ter efeitos negativos para a célula; o ARN modificado de maneira incompleta que chega ao citoplasma é degradado em vez de ser utilizado na tradução em proteínas.[4]
Agregação e desagregação |
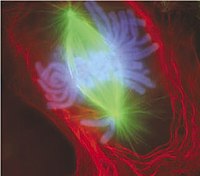
Imagem de célula do pulmão de um tritão, durante a metáfase, na qual foi aplicado um corante fluorescente. O aparelho mitótico pode ser visto, corado a verde, agregado aos dois conjuntos de cromossomas que estão corados a azul. Todos os cromossomas menos um estão na placa metafásica.
Durante o seu ciclo de vida, o núcleo pode se desagregar, quer em resposta ao processo de divisão celular quer como consequência da apoptose, uma forma de morte celular programada. Durante estes eventos, os componentes estruturais do núcleo, o envelope e a lâmina, são sistematicamente degradados.
Durante o ciclo celular, a célula divide-se para formar duas células. Para que este processo seja possível, cada uma das células resultantes deverá possuir um conjunto completo de genes, um processo que requer a replicação dos cromossomas, assim como a segregação em conjuntos separados. Isto ocorre pelos cromossomas replicados, os cromatídeos irmãos, ligados aos microtúbulos, que por sua vez estão ligados a diferentes centrossomas. Os cromatídeos irmãos podem então ser puxados para diferentes localizações na célula. No entanto, em muitas células, o centrossoma está localizado no citoplasma, fora do núcleo, e os microtúbulos não podem ligar-se aos cromatídeos na presença de um envelope nuclear.[42] Portanto, nos passos iniciais do ciclo celular, começando na prófase até cerca da prometáfase, a membrana nuclear é desmantelada.[8] Durante o mesmo período, a lâmina nuclear também é desagregada através de um processo regulado por fosforilação das laminas.[43] Para o fim do ciclo celular, a membrana nuclear é novamente agregada, e pela mesma altura a lâmina nuclear também o é, através da desfosforilação das laminas.[43]
A apoptose é um processo controlado, através do qual os componentes estruturais da célula são destruídos, resultando na morte da célula. As mudanças associadas com a apoptose afectam diretamente o núcleo e o seu conteúdo, por exemplo, na condensação da cromatina e desintegração do envelope e lâmina nucleares. A destruição da rede de laminas e controlada por proteases especializadas, denominadas caspases, que fazem a clivagem das laminas, comprometendo dessa forma a integridade estrutural do núcleo. A clivagem das laminas é por vezes usada como um indicador laboratorial da atividade de caspases, em ensaios de atividade precoce de apoptose.[8] Células que expressam laminas resistentes a caspases são deficientes nas mudanças nucleares relacionadas com a apoptose, sugerindo que as laminas desempenham um papel essencial no início dos eventos que levam à degradação do núcleo por apoptose.[8] A própria inibição da agregação das laminas é um indutor da apoptose.[44]
O envelope nuclear age como uma barreira que previne que vírus de ADN e ARN entrem no núcleo. Alguns vírus requerem acesso a proteínas que existem dentro do núcleo de maneira a poderem-se replicar ou agregarem os seus componentes. Os vírus de ADN, como o herpes-vírus, replicam e agregam-se no núcleo celular, saindo depois por evaginação através da membrana nuclear interna. Este processo é acompanhado pela desagregação da lâmina da face nuclear da membrana interna.[8]
Células anucleadas e polinucleadas |
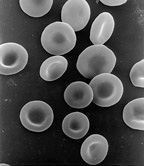
Os eritrócitos humanos, tal como os de outros mamíferos, carecem de núcleo. Este facto faz parte do desenvolvimento normal da célula.
Apesar de a maioria das células possuir um único núcleo, alguns tipos de células não possuem núcleo e outros possuem vários núcleos. Isto pode ser derivado de processos normais, como o da maturação dos eritrócito de mamíferos, ou ser resultado de divisões celulares mal sucedidas.
As células anucleadas não possuem núcleo e portanto são incapazes de se dividirem para produção de descendência celular. O tipo de célula anucleada mais conhecida é o eritrócito de mamíferos, que também carece de outros organelos como a mitocôndria e serve principalmente para o transporte de oxigénio dos pulmões para os tecidos celulares. Os eritrócitos sofrem maturação através do processo denominado eritropoiese, que se dá na medula óssea e onde perdem o núcleo, organelos e ribossomas. O núcleo é expelido durante o processo de diferenciação de um eritroblasto em um reticulócito, o precursor imediato dos eritrócitos maduros.[45] A presença de um agente mutagénicos poderá induzir a libertação de alguns eritrócitos "micronucleados" imaturos.[46][47] Células anucleadas também podem surgir de divisões celulares mal processadas, em que uma das células-filhas não possui núcleo e a outra fica binucleada.
As células polinucleadas possuem múltiplos núcleos. A maioria das espécies de protozoário da classe Acantharea[48] e alguns fungos em micorrizas[49] possuem células polinucleadas. Em humanos, as células do músculo esquelético, denominadas miócitos, tornam-se multinucleadas durante o seu desenvolvimento; o arranjo de núcleos resultante, perto da periferia das células, permite um máximo de espaço intracelular para as miofibrilhas.[4] Células multinucleadas também podem ser anormais em humanos; por exemplo, células que derivam da fusão de monócitos e macrófagos, conhecidas como células gigantes multinucleadas, por vezes acompanham reacções de inflamação[50] e também estão envolvidas na formação de tumores.[51]
Evolução |
Sendo a principal característica que define uma célula eucariótica, a origem evolutiva do núcleo tem sido alvo de muitas especulações. Quatro grandes teorias foram propostas para explicar a existência do núcleo, apesar de nenhuma ter até agora um apoio alargado.[52]
A teoria conhecida como modelo sintrófico propõe que uma relação simbiótica entre as Archaea e as Bacteria terá criado a célula eucariótica portadora de núcleo. Formula-se que a simbiose se originou quando Archaea primitivas, similares às atuais Archaea metanogénicas, invadiram e passaram a viver dentro de bactérias similares às atuais mixobactérias, eventualmente formando um núcleo primordial. Esta teoria é análoga à teoria aceite sobre a origem da mitocôndria eucariótica e do cloroplasto, que se pensa terem se desenvolvido a partir de uma similar relação endossimbiótica entre um proto-eucariotas e bactérias aeróbias.[53] A origem do núcleo entre as Archaea é suportado por observações de que este grupo e os eucariotas possuem genes similares para determinadas proteínas, incluindo as histonas. As observação que mostram as mixobactérias como organismos móveis, que podem formar complexos multicelulares e que possuem quinases e proteínas G similares aos Eukarya, suportam uma origem bacteriana da célula eucariótica.[54]
Um segundo modelo propõe que células proto-eucarióticas evoluíram a partir de bactérias, sem estágios endossimbióticos. Este modelo é baseado na existência das bactérias do filo Planctomycetes, que possuem uma estrutura nuclear com poros primitivos e outras estruturas membranares compartimentadas.[55] Um modelo similar propões que uma célula semelhante à eucariótica, o cronócito, evoluiu primeiramente, tendo depois fagocitado membros das Archaea e Bacteria, gerando assim o núcleo e a célula eucariótica.[56]
O modelo mais controverso, conhecido como eucariogénese viral, propõe que o núcleo composto de membranas, assim como outras estruturas eucarióticas, originaram-se a partir da infecção de um vírus. A sugestão é suportada por similaridades entre eucariotas e vírus: fitas lineares de ADN e ligação forte a proteínas (analogia entre histonas e envelope viral). Uma versão da proposta sugere que o núcleo evoluiu ao mesmo tempo em que a fagocitose, formando um predador celular primitivo.[57] Outra variante propõe que os eucariotas são originários de Archaea primitivos, infectados com poxvirus, baseada nas semelhanças entre a polimerase de ADN de modernos poxvirus e eucariotas.[58][59] Tem sido sugerido que a questão ainda não resolvida da evolução do sexo possa estar ligada à hipótese da eucariogénese viral.[60]
Finalmente, uma proposta recente sugere que variantes tradicionais da teoria da endossimbiose são insuficientemente robustas para explicar a origem do núcleo eucariótico. Este modelo, denominado "hipótese exomembranar", sugere que o núcleo se originou de uma única célula ancestral que formou uma segunda membrana celular externa; a membrana interior que envolvia a célula original tornar-se-ia na membrana nuclear, formando poros mais complexos ao longo do tempo, permitindo a passagem de componentes celulares sintetizados internamente como as subunidades ribossomais
Referências
↑ H Harris (1999). The Birth of the Cell. New Haven: Yale University Press
↑ Brown, Robert (1866). «On the Organs and Mode of Fecundation of Orchidex and Asclepiadea». Miscellaneous Botanical Works. I: 511–514
↑ ab Thomas Cremer (1985). Von der Zellenlehre zur Chromosomentheorie. Berlin, Heidelberg, New York, Tokyo: Springer Verlag. ISBN 3-540-13987-7 versão online here
↑ abcdefgh H Lodish; Berk A, Matsudaira P, Kaiser CA, Krieger M, Scott MP, Zipursky SL, Darnell J. (2004). Molecular Cell Biology 5th ed. New York: WH Freeman A referência emprega parâmetros obsoletos|coautores=(ajuda)
↑ abcd Bruce Alberts, Alexander Johnson, Julian Lewis, Martin Raff, Keith Roberts, Peter Walter, ed. (2002). Molecular Biology of the Cell 4th ed. [S.l.]: Garland Science !CS1 manut: Nomes múltiplos: lista de editores (link)
↑ Stuurman N, Heins S, Aebi U (1998). «Nuclear lamins: their structure, assembly, and interactions». J Struct Biol. 122 (1–2): 42–66. PMID 9724605 !CS1 manut: Nomes múltiplos: lista de autores (link)
↑ Goldman A, Moir R, Montag-Lowy M, Stewart M, Goldman R (1992). «Pathway of incorporation of microinjected lamin A into the nuclear envelope». J Cell Biol. 119 (4): 725–735. PMID 1429833 !CS1 manut: Nomes múltiplos: lista de autores (link)
↑ abcde Goldman R, Gruenbaum Y, Moir R, Shumaker D, Spann T (2002). «Nuclear lamins: building blocks of nuclear architecture». Genes Dev. 16 (5): 533–547. PMID 11877373 !CS1 manut: Nomes múltiplos: lista de autores (link)
↑ Moir RD, Yoona M, Khuona S, Goldman RD. (2000). «Nuclear Lamins A and B1: Different Pathways of Assembly during Nuclear Envelope Formation in Living Cells». Journal of Cell Biology. 151 (6): 1155–1168. PMID 11121432 !CS1 manut: Nomes múltiplos: lista de autores (link)
↑ Spann TP, Goldman AE, Wang C, Huang S, Goldman RD. (2002). «Alteration of nuclear lamin organization inhibits RNA polymerase II–dependent transcription». Journal of Cell Biology. 156 (4): 603–608. PMID 11854306 !CS1 manut: Usa parâmetro autores (link)
↑ Ehrenhofer-Murray A (2004). «Chromatin dynamics at DNA replication, transcription and repair». Eur J Biochem. 271 (12): 2335–2349. PMID 15182349
↑ Grigoryev S, Bulynko Y, Popova E (2006). «The end adjusts the means: heterochromatin remodelling during terminal cell differentiation». Chromosome Res. 14 (1): 53–69. PMID 16506096 !CS1 manut: Nomes múltiplos: lista de autores (link)
↑ Schardin, Margit; T. Cremer, H. D. Hager, M. Lang (December 1985). «Specific staining of human chromosomes in Chinese hamster x man hybrid cell lines demonstrates interphase chromosome territories». Springer Berlin / Heidelberg. Human Genetics. 71 (4): 281–287. doi:10.1007/BF00388452. PMID 2416668 A referência emprega parâmetros obsoletos|coautores=(ajuda); Verifique data em:|data=(ajuda)
↑ Lamond, Angus I.; William C. Earnshaw (24 April 1998). «Structure and Function in the Nucleus». Science. 280: 547–553. PMID 9554838 A referência emprega parâmetros obsoletos|coautores=(ajuda); Verifique data em:|data=(ajuda)
↑ Kurz, A; S Lampel, JE Nickolenko, J Bradl, A Benner, RM Zirbel, T Cremer and P Lichter (1996). «Active and inactive genes localize preferentially in the periphery of chromosome territories». The Rockefeller University Press. The Journal of Cell Biology. 135: 1195–1205. PMID 8947544 A referência emprega parâmetros obsoletos|coautores=(ajuda)
↑ NF Rothfield, BD Stollar (1967). «The Relation of Immunoglobulin Class, Pattern of Antinuclear Antibody, and Complement-Fixing Antibodies to DNA in Sera from Patients with Systemic Lupus Erythematosus». J Clin Invest. 46 (11): 1785–1794. PMID 4168731
↑ S Barned, AD Goodman, DH Mattson (1995). «Frequency of anti-nuclear antibodies in multiple sclerosis». Neurology. 45 (2): 384–385. PMID 7854544 !CS1 manut: Nomes múltiplos: lista de autores (link)
↑ Paine P, Moore L, Horowitz S (1975). «Nuclear envelope permeability». Nature. 254 (5496): 109–114. PMID 1117994 !CS1 manut: Nomes múltiplos: lista de autores (link)
↑ Rodney Rhoades, Richard Pflanzer, ed. (1996). «Ch3». Human Physiology 3rd ed. [S.l.]: Saunders College Publishing
↑ Shulga N, Mosammaparast N, Wozniak R, Goldfarb D (2000). «Yeast nucleoporins involved in passive nuclear envelope permeability». J Cell Biol. 149 (5): 1027–1038. PMID 10831607 !CS1 manut: Nomes múltiplos: lista de autores (link)
↑ ab Pemberton L, Paschal B (2005). «Mechanisms of receptor-mediated nuclear import and nuclear export». Traffic. 6 (3): 187–198. PMID 15702987
↑ Hernandez-Verdun, Daniele (2006). «Nucleolus: from structure to dynamics». Histochem. Cell. Biol (125): 127–137. doi:10.1007/s00418-005-0046-4
↑ ab Lamond, Angus I.; Judith E. Sleeman. «Nuclear substructure and dynamics». Current Biology. 13 (21): R825–828. PMID 14588256 A referência emprega parâmetros obsoletos|coautores=(ajuda)
↑ abc Cioce M, Lamond A. «Cajal bodies: a long history of discovery». Annu Rev Cell Dev Biol. 21: 105–131. PMID 16212489
↑ abc Thomas D. Pollard; William C. Earnshaw (2004). Cell Biology. Philadelphia: Saunders. ISBN 0-7216-3360-9 A referência emprega parâmetros obsoletos|coautores=(ajuda)
↑ abc Dundr, Miroslav; Tom Misteli (2001). «Functional architecture in the cell nucleus». Biochem. J. (356): 297–310. PMID 11368755 A referência emprega parâmetros obsoletos|coautores=(ajuda)
↑ Fox, Archa (7 de março de 2007). «Paraspeckle Size» (entrevista). Entrevista com R. Sundby. R. Sundby. E-mail Correspondence
↑ abc Matera AG, Frey MA. (1998). «Coiled Bodies and Gems: Janus or Gemini?». American Journal of Human Genetics. 63 (2): 317–321. PMID 9683623
↑ Matera, A. Gregory (1998). «Of Coiled Bodies, Gems, and Salmon». Journal of Cellular Biochemistry (70): 181–192. PMID 9671224
↑ Saunders WS, Cooke CA, Earnshaw WC (1991). «Compartmentalization within the nucleus: discovery of a novel subnuclear region.». Journal of Cellular Biology. 115 (4): 919–931 !CS1 manut: Nomes múltiplos: lista de autores (link) PMID 1955462
↑ Pombo A, Cuello P, Schul W, Yoon J, Roeder R, Cook P, Murphy S (1998). «Regional and temporal specialization in the nucleus: a transcriptionally active nuclear domain rich in PTF, Oct1 and PIKA antigens associates with specific chromosomes early in the cell cycle». EMBO J. 17 (6): 1768–1778. PMID 9501098 !CS1 manut: Nomes múltiplos: lista de autores (link)
↑ ab Fox, Archa; et al. (2002). = PIIS0960982201006327 «Paraspeckles:A Novel Nuclear Domain» Verifique valor|url=(ajuda). Current Biology. 12: 13–25 !CS1 manut: Uso explícito de et al. (link)
↑ Fox, Archa; Wendy Bickmore (2004). «Nuclear Compartments: Paraspeckles». Nuclear Protein Database. Consultado em 6 de março de 2007 A referência emprega parâmetros obsoletos|coautor=(ajuda)
↑ ab Fox, A.; et al. (2005). «P54nrb Forms a Heterodimer with PSP1 That Localizes to Paraspeckles in an RNA-dependent Manner». Molecular Biology of the Cell. 16: 5304–5315 !CS1 manut: Uso explícito de et al. (link) PMID 16148043
↑ Handwerger, Korie E.; Joseph G. Gall (January 2006). «Subnuclear organelles: new insights into form and function». TRENDS in Cell Biology. 16 (1): 19–26. doi:10.1016/j.tcb.2005.11.005 A referência emprega parâmetros obsoletos|coautores=(ajuda); Verifique data em:|data=(ajuda)
↑ Albert L. Lehninger; David L. Nelson, Michael M. Cox. (2000). Lehninger principles of biochemistry 3rd ed. New York: Worth Publishers. ISBN 1-57259-931-6 A referência emprega parâmetros obsoletos|coautores=(ajuda)
↑ Moreno F, Ahuatzi D, Riera A, Palomino CA, Herrero P. (2005). «Glucose sensing through the Hxk2-dependent signalling pathway.». Biochem Soc Trans. 33 (1): 265–268 !CS1 manut: Nomes múltiplos: lista de autores (link) PMID 15667322
↑ Görlich, Dirk; Ulrike Kutay (1999). «Transport between the cell nucleus and the cytoplasm». Ann. Rev. Cell Dev. Biol. (15): 607–660. PMID 10611974 A referência emprega parâmetros obsoletos|coautores=(ajuda)
↑ Knud H. Nierhaus; Daniel N. Wilson (2004). Protein Synthesis and Ribosome Structure: Translating the Genome. [S.l.]: Wiley-VCH. ISBN 3-527-30638-2 A referência emprega parâmetros obsoletos|coautores=(ajuda)
↑ Claudio A. Nicolini (1997). Genome Structure and Function: From Chromosomes Characterization to Genes Technology. [S.l.]: Springer. ISBN 0-7923-4565-7
↑ JD Watson; Baker TA, Bell SP, Gann A, Levine M, Losick R. (2004). «Ch9–10». Molecular Biology of the Gene 5th ed. [S.l.]: Peason Benjamin Cummings; CSHL Press. A referência emprega parâmetros obsoletos|coautores=(ajuda)
↑ Lippincott-Schwartz, Jennifer (7 March 2002). «Cell biology: Ripping up the nuclear envelope». Nature. 416 (6876): 31–32. doi:10.1038/416031a Verifique data em:|data=(ajuda)
↑ ab Boulikas T (1995). «Phosphorylation of transcription factors and control of the cell cycle». Crit Rev Eukaryot Gene Expr. 5 (1): 1–77. PMID 7549180
↑ Steen R, Collas P (2001). «Mistargeting of B-type lamins at the end of mitosis: implications on cell survival and regulation of lamins A/C expression». J Cell Biol. 153 (3): 621–626. PMID 11331311
↑ Skutelsky, E.; Danon D. (June 1970). «Comparative study of nuclear expulsion from the late erythroblast and cytokinesis». J Cell Biol (60(3)): 625–635. PMID 5422968 A referência emprega parâmetros obsoletos|coautores=(ajuda); Verifique data em:|data=(ajuda)
↑ Torous, DK; Dertinger SD, Hall NE, Tometsko CR. (2000). «Enumeration of micronucleated reticulocytes in rat peripheral blood: a flow cytometric study». Mutat Res (465(1–2)): 91–99. PMID 10708974 A referência emprega parâmetros obsoletos|coautores=(ajuda)
↑ Hutter, KJ; Stohr M. (1982). «Rapid detection of mutagen induced micronucleated erythrocytes by flow cytometry». Histochemistry (75(3)): 353–362. PMID 7141888 A referência emprega parâmetros obsoletos|coautores=(ajuda)
↑ Zettler, LA; Sogin ML, Caron DA (1997). «Phylogenetic relationships between the Acantharea and the Polycystinea: A molecular perspective on Haeckel's Radiolaria». Proc Natl Acad Sci USA (94): 11411–11416. PMID 9326623 A referência emprega parâmetros obsoletos|coautores=(ajuda)
↑ Horton, TR (2006). «The number of nuclei in basidiospores of 63 species of ectomycorrhizal Homobasidiomycetes». Mycologia (98(2)): 233–238. PMID 16894968
↑ McInnes, A; Rennick DM (1988). «Interleukin 4 induces cultured monocytes/macrophages to form giant multinucleated cells». J Exp Med (167): 598–611. PMID 3258008 A referência emprega parâmetros obsoletos|coautores=(ajuda)
↑ Goldring, SR; Roelke MS, Petrison KK, Bhan AK (1987). «Human giant cell tumors of bone identification and characterization of cell types». J Clin Invest (79(2)): 483–491. PMID 3027126 A referência emprega parâmetros obsoletos|coautores=(ajuda)
↑ Pennisi E. (2004). «Evolutionary biology. The birth of the nucleus». Science. 305 (5685): 766–768. PMID 15297641
↑ Margulis, Lynn (1981). Symbiosis in Cell Evolution. San Francisco: W. H. Freeman and Company. pp. 206–227. ISBN 0-7167-1256-3
↑ Lopez-Garcia P, Moreira D. (2006). «Selective forces for the origin of the eukaryotic nucleus». Bioessays. 28 (5): 525–533. PMID 16615090
↑ Fuerst JA. (2005). «Intracellular compartmentation in planctomycetes». Annu Rev Microbiol. 59: 299–328. PMID 15910279
↑ Hartman H, Fedorov A. (2002). «The origin of the eukaryotic cell: a genomic investigation». Proc Natl Acad Sci U S A. 99 (3): 1420–1425. PMID 11805300
↑ Bell PJ. (2001). "Viral eukaryogenesis: was the ancestor of the nucleus a complex DNA virus?" J Mol Biol Sep;53(3):251–256. PMID 11523012
↑ Takemura M. (2001). Poxviruses and the origin of the eukaryotic nucleus. J Mol Evol 52(5):419–425. PMID 11443345
↑ Villarreal L, DeFilippis V (2000). «A hypothesis for DNA viruses as the origin of eukaryotic replication proteins». J Virol. 74 (15): 7079–7084. PMID 10888648
↑ Bell PJ. (2006). "Sex and the eukaryotic cell cycle is consistent with a viral ancestry for the eukaryotic nucleus." J Theor Biol 2006 November 7;243(1):54–63. PMID 16846615
Leitura adicional |
Goldman, Robert D.; Yosef Gruenbaum, Robert D. Moir, Dale K. Shumaker and Timothy P. Spann (2002). «Nuclear lamins: building blocks of nuclear architecture». Genes & Dev. (16): 533–547. doi:10.1101/gad.960502 A referência emprega parâmetros obsoletos|coautores=(ajuda)
Görlich, Dirk; Ulrike Kutay (1999). «Transport between the cell nucleus and the cytoplasm». Ann. Rev. Cell Dev. Biol. (15): 607–660. PMID 10611974 A referência emprega parâmetros obsoletos|coautores=(ajuda)
Lamond, Angus I.; William C. Earnshaw (24 APRIL 1998). «Structure and Function in the Nucleus». Science. 280: 547–553. PMID 9554838 A referência emprega parâmetros obsoletos|coautores=(ajuda); Verifique data em:|data=(ajuda)
Pennisi E. (2004). «Evolutionary biology. The birth of the nucleus». Science. 305 (5685): 766–768. doi:10.1126/science.305.5685.766. PMID 15297641
Ligações externas |
Dicas Núcleo Celular (em português) Atividades práticas para a compreensão do núcleo celular.- (em português) Sem Nome. Uma Organela no Núcleo Celular (pdf) Notícia sobre uma nova organela celular localizada dentro do núcleo.
cellnucleus.com (em inglês) Sítio que cobre a temática da estrutura e função do Núcleo - Departamento de Oncologia da Universidade de Alberta
The Nuclear Protein Database Informações sobre componentes nucleares. (em inglês)
The Nucleus Collection (em inglês) - Image & Video Library - The American Society for Cell Biology Contém imagens e vídeos que ilustram o núcleo
Nuclear Envelope and Nuclear Import Section - Landmark Papers in Cell Biology, Joseph G. Gall, J. Richard McIntosh, eds., contém comentários e ligações para artigos sobre o núcleo. Publicado online em Image & Video Library - The American Society for Cell Biology (em inglês)
