Glicose-6-fosfato
| Glicose-6-fosfato Alerta sobre risco à saúde | |
|---|---|
 |  |
| Outros nomes | Éster de Robinson, G-6-P |
| Identificadores | |
Número CAS | , 5996-17-8 (sal de potássio) |
PubChem | |
MeSH | |
SMILES |
|
| Propriedades | |
Fórmula molecular | C6H11O9P |
Massa molar | 260.136 |
| Riscos associados | |
Frases R | R34 |
Frases S | S26 S27 S36/37/39 S45 |
Exceto onde denotado, os dados referem-se a materiais sob condições normais de temperatura e pressão Referências e avisos gerais sobre esta caixa. Alerta sobre risco à saúde. | |

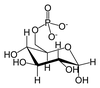
Estrutura da glicose 6-fosfato mostrada utilizando projeção de Fischer, à esquerda, e modelo poligonal [1], à direita.
A glicose-6-fosfato é um açúcar (glicose) fosforilado no carbono nº 6. Este composto é muito comum nas células: a vasta maioria da glicose que entra na célula fica fosforilada desta forma.
Devido à sua proeminente posição na química celular, a glicose-6-fosfato possui destinos muito variados dentro da célula. Pertence ao início de duas das maiores vias metabólicas:
- Glicólise
- Via das pentoses-fosfato
Em adição a estas vias metabólicas, a glicose-6-fosfato também pode ser convertida em glicogénio ou amido para armazenamento. Este armazenamento dá-se no fígado e nos músculos, sob a forma de glicogénio para a maioria dos animais multicelulares e de amido intracelular ou grânulos de glicogénio para a maioria do resto dos organismos.
Ver também |
- Glicose
- Glicogénio
- Glicose-1-fosfato
- Via das pentoses-fosfato
Deficiência em glucose-6-fosfato desidrogenase
↑ Bonafe, C. F. S.; Bispo, J. A. C.; de Jesus, M. B. (2018). The Polygonal Model: A Simple Representation of Biomolecules as a Tool for Teaching Metabolism. Biochemistry and Molecular Biology Education. 46: 66-75. DOI - 10.1002/bmb.21093.